- Le Laboratoire
- Biologie moléculaire
- Module logiciel d'analyse
- JSI medical systems

- Produits
- Catalogues
- News & Trends
- Salons
Module logiciel pour séquençage NGS SEQNEXTd'analysede capturede cartographie
Ajouter à mes favoris
Ajouter au comparateur
Vous voulez acheter directement ?
Rendez-vous sur notre Shop.
Caractéristiques
- Fonction
- d'analyse, de capture, de cartographie
- Applications
- de laboratoire, pour séquençage NGS
Description
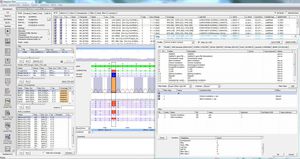
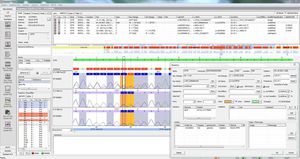
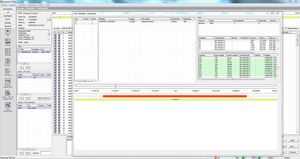
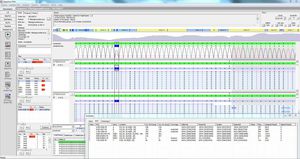
SEQNEXT est une application puissante et conviviale pour la cartographie, l'alignement et la détection de variantes des données de séquençage de nouvelle génération. SEQNEXT peut analyser des données provenant de toutes les plateformes et kits de séquençage courants (basés sur la PCR, l'enrichissement, la capture de séquences). La visualisation de tous les variants détectés, tels que les délétions, les insertions et les indels de toute longueur, les SNP, les régions répétées ainsi que les CNV et les fusions de gènes, est claire et intuitive.
SEQNEXT offre un accès aux bases de données SNP publiques telles que dbSNP, 1000 Genomes, ClinVar, ExAC et gnomAD pour la classification et le filtrage. Toutes les données de résultats peuvent être échangées avec notre base de données de variantes pour l'expérience et la connaissance partagées - varSEAK, transférées vers les systèmes LIM internes des laboratoires et / ou émises sous forme de rapports personnalisés pour les patients.
NOUVEAU Analyse améliorée des données de séquençage de virus, par exemple SARS-CoV-2
Trioanalyse et Poolanalyse améliorée
Annotations de l'ontologie de la séquence dans le tableau des variantes
Option de saut vers varSEAK et toutes les bases de données externes disponibles telles que dbSNP, ClinVar, UK10K, ESP, gnomAD et ExAC
Analyse du séquençage de l'exome entier (WES)
Compatible avec les données provenant de toutes les plateformes et kits de séquençage de nouvelle génération courants
Mise en place facile de régions cibles individuelles (ROIs) via l'importation de fichiers bed- ou manifest basés sur hg19 et/ou hg38
Importation de listes de gènes pour le filtrage et l'analyse des données WES
Analyse des fichiers fastq, fastq.gz ou bam
Normes efficaces pour la cartographie, l'alignement, la qualité et l'appel de variants (algorithmes utilisés : BWA et Smith-Waterman, adaptés par JSI)
Paramètres personnalisés pour l'analyse et l'appel de variants (≥ 0,1 %)
Sensibilité et spécificité élevées pour la détection des délétions, insertions et indels de toute longueur, des SNP, des régions répétées ainsi que des CNV et des fusions de gènes
---
Catalogues
SEQUENCE PILOT
2 Pages
* Les prix s'entendent hors taxe, hors frais de livraison, hors droits de douane, et ne comprennent pas l'ensemble des coûts supplémentaires liés aux options d'installation ou de mise en service. Les prix sont donnés à titre indicatif et peuvent évoluer en fonction des pays, des cours des matières premières et des taux de change.